Zur Übergabe der Sequenzdaten und Parameter wird eine Eingabedatei
verwendet.
Die Eingabedatei entspricht dem normalerweise bei PHILIP-Programmen
verwendeten Format (Felsenstein, 1993).
In der einfachsten Form enthält sie nur alignierte Sequenzen
verschiedener Organismen sowie deren Anzahl und die Zahl der Spalten
des Alignments. Diese können in einem
,,interleaved``![[*]](foot_motif.gif) oder sequentiellen Format vorliegen.
Am Beginn der ersten Zeile findet man immer zwei Zahlen, die die
Anzahl der benutzten Spezies und an zweiter Stelle die Anzahl der
Spalten im Alignment angeben.
Danach folgen die Sequenzen. Die ersten zehn Buchstaben enthalten
den Namen der Spezies oder Sequenz, alle darauffolgenden Zeilen
enthalten die Sequenz, bei der nur Buchstaben, Punkte, Fragezeichen
und Striche verwendet werden. Ziffern und alle Arten von Leerzeichen
(wie z.B. Tabstops) werden ignoriert.
Im ,,interleaved`` Format, dem Standardformat, das der Ausgabe
vieler Alignmentprogramme gleicht, wird jeweils eine Zeile
für jede Spezies in Blöcken nacheinander abgelegt.
oder sequentiellen Format vorliegen.
Am Beginn der ersten Zeile findet man immer zwei Zahlen, die die
Anzahl der benutzten Spezies und an zweiter Stelle die Anzahl der
Spalten im Alignment angeben.
Danach folgen die Sequenzen. Die ersten zehn Buchstaben enthalten
den Namen der Spezies oder Sequenz, alle darauffolgenden Zeilen
enthalten die Sequenz, bei der nur Buchstaben, Punkte, Fragezeichen
und Striche verwendet werden. Ziffern und alle Arten von Leerzeichen
(wie z.B. Tabstops) werden ignoriert.
Im ,,interleaved`` Format, dem Standardformat, das der Ausgabe
vieler Alignmentprogramme gleicht, wird jeweils eine Zeile
für jede Spezies in Blöcken nacheinander abgelegt.
Zwischen den einzelnen Blöcken kann jeweils eine Leerzeile
eingefügt werden.
Im sequentiellen Format folgt nach dem Namen erst die vollständige
Sequenz dieser Spezies, bevor die nächste Spezies aufgelistet
wird:
Neben den Sequenzdaten können noch weitere Parameter und Optionen
nach den Zahlenangaben und vor den Sequenzdaten angegeben werden.
Die Optionen folgen nach den Sequenzdaten auch in der ersten Zeile.
Zu den Optionen gehörige Parameter folgen in den anschließenden
Zeilen.
- 1 - Sequenzdaten ausgeben
Normalerweise werden die Sequenzdaten nicht ausgegeben.
Dieses wird durch diese Option aufgehoben.
- 3 - Keinen Baum mit ausgeben
Bei Angabe dieser Option wird kein Baum nach der
Analyse ausgegeben, was normalerweise der Fall wäre.
- 4 - Baum in eine Datei schreiben
Normalerweise werden Bäume am Ende nicht im Newick-Format
in eine Baumdatei geschrieben, es sei denn, diese Option
wurde angegeben.
- B - Bootstrap-Stichprobe ziehen
Durch diese Option wird veranlaßt, daß pfastDNAml
eine Bootstrap-Stichprobe von den Eingabedaten generiert
und mit dieser Stichprobe die Stammbaumberechnungen
durchführt. Neben dieser Option muß in einer der Zeilen bis zu
den Sequenzdaten ein sogenannte Random-Number-Seed zu
Initialisierung des Zufallsgenerators in der Form
,,B n`` angegeben werden. Bei n handelt es sich
um eine ganze Zahl.
Um unterschiedliche Stichproben zu erhalten, muß jedesmal ein
anderer Random-Number-Seed verwendet werden;
d.h. bei gleichen Werten für n ist auch die gezogene
Stichprobe dieselbe. Beispiel:
5 42 B
B 127
- C - Kategorien anlegen
Mit dieser Option ist es möglich, jede Spalte im Alignment
einer Kategorie zuzuordnen. Hierzu stehen bis zu 35 Kategorien
(1, 2, ..., 9, A, B, ..., Y, Z) zur Verfügung.
Jeder der benutzten Kategorien muß ein Gewicht zugeordnet werden,
das eine Dezimalzahl sein kann. Diese Gewichte werden in einer
Zeile der Form ,,C 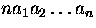 `` angegeben.
n ist die Anzahl der benutzten Kategorien und a1 bis an
die vergebenen Gewichte (a1 ist das Gewicht für Kategorie 1,
a2 für Kategorie 2 usw.). In einer separaten Zeile, die
mit dem Schlüsselwort Categories beginnt, wird für jede
Spalte im Alignment eine Kategorie vergeben.
Beispiel:
`` angegeben.
n ist die Anzahl der benutzten Kategorien und a1 bis an
die vergebenen Gewichte (a1 ist das Gewicht für Kategorie 1,
a2 für Kategorie 2 usw.). In einer separaten Zeile, die
mit dem Schlüsselwort Categories beginnt, wird für jede
Spalte im Alignment eine Kategorie vergeben.
Beispiel:
5 42 C
C 12 0.0625 0.125 0.25 0.5 1 2 4 8 16 32 64 128
Categories 51388923A11555238BBAAA112348973621123789AB
- F - Empirische Basenfrequenzen
Diese Option bringt pfastDNAml dazu, die Frequenzen der
organischen Basen anhand der übergebenen Sequenzdaten zu berechnen.
Wird diese Option nicht gesetzt, müssen die Basenfrequenzen in
einer eigenen Zeile vor den Sequenzdaten übergeben werden.
Beispiel für gleichverteilte Basen:
0.25 0.25 0.25 0.25
- G - Globale Rearrangements
Diese Option sorgt dafür, daß die Werte gesetzt werden können,
über wieviel Äste hinweg die ausgeführten Rearrangements
(siehe Seite ![[*]](cross_ref_motif.gif) ) durchgeführt werden sollen.
) durchgeführt werden sollen.
Hierzu kann optional eine Parameterzeile ,,G n1 n2``
angegeben werden, in der n2 angibt, über wieviele Äste die
Rearrangements während des Einfügens gehen sollen, und n1
die Anzahl für das Rearrangement am Ende der Analyse.
Hierbei muß 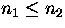 gelten.
gelten.
Wird n2 nicht angegeben, wird der Standardwert 1 verwendet.
Ist die Parameterzeile gar nicht gesetzt, wird am Ende
ein globales Rearrangement durchgeführt.
- I - Sequentielles Datenformat (not interleaved)
Diese Option gibt an, daß die Sequenzdaten im sequentiellen
Format vorliegen.
- J - Speziesreihenfolge mischen (Jumble)
Bei Angabe dieser Option werden die Spezies in zufälliger
Reihenfolge in die bestehenden Bäume eingefügt.
Zur Initialisierung des Zufallsgenerators muß auch hier,
wie bei Bootstrap-Analysen, ein Random-Number-Seed übergeben
werden. Beispiel:
5 42 J
J 1357
- O - Außengruppe benutzen (Outgroup)
Über die dazugehörige Parameterzeile wird angegeben,
mit welcher Spezies als Außengruppe der berechnete
Baum gewurzelt werden soll.
Beispiel mit Spezies 3 als Außengruppe:
5 42 O
O 3
- Q - Schnelles Einfügen (Quickadd)
Diese Option beschleunigt die Baumkonstruktion dadurch,
daß beim Einfügen neuer Spezies nicht alle, sondern nur
die unmittelbar betroffenen Kanten optimiert werden.
- R - Neustart (Restart)
Mit dieser Option kann pfastDNAml, z.B. nach einem
Abbruch, erneut gestartet werden.
Hierzu muß am Ende der Eingabedatei der Baum, nach dem
die vorherige Berechnung abgebrochen wurde, nach den
Sequenzdaten angefügt werden.
Diesen Baum findet man am Ende der checkpoint-Datei
des abgebrochenen Jobs. Von diesem muß vor dem Neustart
noch der Kommentar entfernt werden.
- T - Transition/Transversions-Rate eingeben
Zusammen mit dieser Option muß in einer Parameterzeile
,,T m`` die Transition/Transversions-Rate m
eingegeben werden. Die Grundeinstellung, wenn diese Option nicht
angegeben wird, ist ein Wert von 2,0.
- W - Gewichte eingeben
Nach der W-Option kann man für jede Spalte
im Alignment ein ganzzahliges Gewicht zwischen und 35
angeben.
Diese Gewichte geben an, wie oft eine Spalte im Alignment
in der Berechnung des Likelihoodwertes berücksichtigt werden
soll. Auf diese Weise können auch von außen Bootstrap-Stichproben
eingegeben werden, ohne daß die Sequenzdaten neu gemischt
werden müßten.
Die Gewichte werden über eine oder mehrere aufeinanderfolgende
Parameterzeilen an das Programm übergeben.
Beispiel:
5 42 W
Weights 11111000101000011111
1100101000011111111001
- Y - Baum in eine Datei schreiben
Schreibt den entgültigen Baum in eine Baumdatei.
Standardmäßig passiert dies im Newick-Format,
mit der Angabe der Parameterzeile ,,Y 2``
geschieht diese im Prolog-Format.
Bei den optionalen Parameterzeilen ist zu beachten, daß sie nicht
als letzte vor den Sequenzdaten stehen dürfen, da sie dann vom
Programm eventuell nicht erkannt werden.
Es kann mit der T-Option und der dann obligatorischen
Parameterzeile ,,T 2.0`` oder einer anderen Rate,
falls bekannt, vor den Sequenzdaten Abhilfe geschaffen werden.
![[*]](foot_motif.gif) oder sequentiellen Format vorliegen.
Am Beginn der ersten Zeile findet man immer zwei Zahlen, die die
Anzahl der benutzten Spezies und an zweiter Stelle die Anzahl der
Spalten im Alignment angeben.
Danach folgen die Sequenzen. Die ersten zehn Buchstaben enthalten
den Namen der Spezies oder Sequenz, alle darauffolgenden Zeilen
enthalten die Sequenz, bei der nur Buchstaben, Punkte, Fragezeichen
und Striche verwendet werden. Ziffern und alle Arten von Leerzeichen
(wie z.B. Tabstops) werden ignoriert.
Im ,,interleaved`` Format, dem Standardformat, das der Ausgabe
vieler Alignmentprogramme gleicht, wird jeweils eine Zeile
für jede Spezies in Blöcken nacheinander abgelegt.
oder sequentiellen Format vorliegen.
Am Beginn der ersten Zeile findet man immer zwei Zahlen, die die
Anzahl der benutzten Spezies und an zweiter Stelle die Anzahl der
Spalten im Alignment angeben.
Danach folgen die Sequenzen. Die ersten zehn Buchstaben enthalten
den Namen der Spezies oder Sequenz, alle darauffolgenden Zeilen
enthalten die Sequenz, bei der nur Buchstaben, Punkte, Fragezeichen
und Striche verwendet werden. Ziffern und alle Arten von Leerzeichen
(wie z.B. Tabstops) werden ignoriert.
Im ,,interleaved`` Format, dem Standardformat, das der Ausgabe
vieler Alignmentprogramme gleicht, wird jeweils eine Zeile
für jede Spezies in Blöcken nacheinander abgelegt.
![[*]](cross_ref_motif.gif)