


Next: Diskussion
Up: Eukaryotenentwicklung
Previous: 18S rRNA
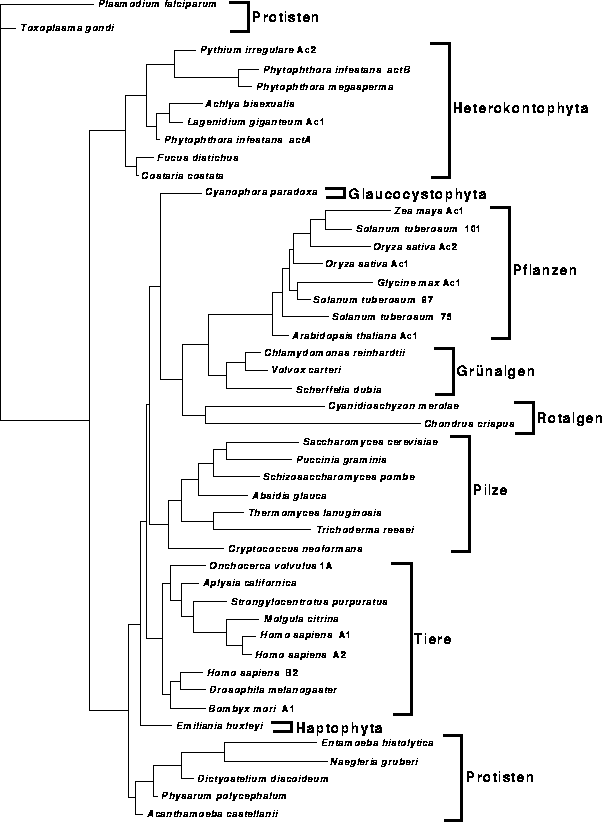
Abbildung:
Phylogenetischer Stammbaum der Eukaryotenentwicklung;
berechnet aus Aktinsequenzen mit pfastDNAml
unter Benutzung der
Codon-Positionen 1 und 2
 |
Neben dem oben beschriebenen 18S rRNA-Datensatz wurde noch
ein zweiter Datensatz aus Aktinsequenzen erstellt, um an
diesem die Nutzbarkeit von Aktin zur Untersuchung der
Plastidenentwicklung zu überprüfen.
Hierfür wurden neben anderen Linien vor allem Vertreter aus
allen bisher schon untersuchten Organismengruppen mit
einfachen Plastiden (Glaucocystophyta, Rotalgen, Grünalgen und Pflanzen)
ausgewählt.
Der benutzte Datensatz enthielt 46 Organismen mit einem Alignment
von 744 Spalten. Auch hier wurden nur die ersten beiden Codon-Positionen
der Aktinsequenzen verwendet. Von diesen 744 Spalten zog
pfastDNAml 394 zur Analyse heran.
Die empirisch ermittelten Basenfrequenzen waren 0,294 (A), 0,221 (C),
0,258 (G) und 0,227 (T). Hieraus ergab sich ein
Transition/Transversions-Parameter von 1,4786.
Nach vier globalen Rearrangement mit je 7310 Bäumen und ingesamt
33764 untersuchten Bäumen wurde ein Stammbaum (Abb. 6.16)
mit Log-Likelihood-Wert -9228,87 gefunden.
Heiko Schmidt
7/17/1997

