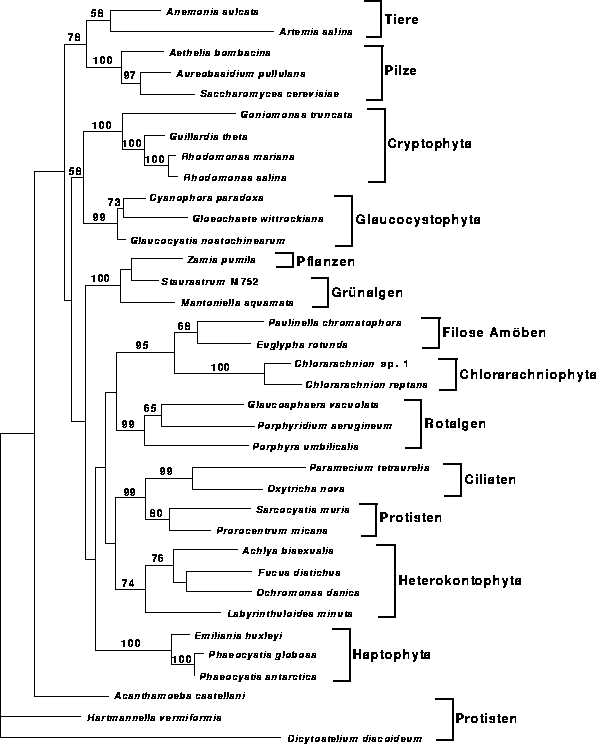
 |
Diese Auswahl geschah so, daß alle in der Untersuchung der Plastidenentwicklung wichtigen Organismenlinien vertreten sind, die Zahl der Organismen aber dennoch eine Bootstrap-Analyse zuläßt. Der erstellte Datensatz enthält 36 Spezies mit einem Alignment über 1565 Sequenzstellen, von denen 802 zur Berechnung des Baumes genutzt wurden.
Bei der Erstellung des Baumes (Abb. 6.15) wurden
11622 Bäume untersucht und zwei globale Rearrangements
mit je 4290 Bäumen durchgeführt.
Es wurden die Basenfrequenzen 0,270 (A), 0,198 (C), 0,265 (G)
und 0,266 (T) und der Transition/Transversions-Parameter 1,5068
empirisch aus den Datenmaterial berechnet.
Der Log-Likelihood-Wert des Baumes ist -21932,35.
 |
Zu dem Baum wurde anschließend eine Bootstrap-Analyse anhand von
100 Pseudostichproben aus dem Datensatz durchgeführt.
Im Konsensusbaum gefundene Bootstrap-Werte über ![]() wurden in Abb. 6.15 an den entsprechenden Kanten
eingefügt.
wurden in Abb. 6.15 an den entsprechenden Kanten
eingefügt.