


Next: Analyse mit gewichtetem Datensatz
Up: Datensatz plast2
Previous: Datensatz plast2
Im zweiten Plastiden-Datensatz wurden die drei Spezies
Corethron criophilum (Heterokontophyta),
Prochlorococcus sp. und
Prochloron didemni (Cyanobakterien) durch andere ersetzt.
Zum einen wurde die Jochalge Chara sp. zugefügt,
die Organisationsformen aufweist, die an Pflanzen erinnern.
Hierdurch sollten die Verwandtschaftsverhältnisse zwischen
höherentwickelten Grünalgen und Pflanzen etwas näher beleuchtet
werden.
Außerdem wurde ein ursprünglicheres Cyanobakterium
Gloeobacter violacius eingefügt, um in den Bereich der
Wurzel des Plastidenastes eine bessere Auflösung zu bringen
und die Auswirkungen auf die berechneten Abzweigungen von Cyanellen
und Chloroplasten zu studieren.
Zusätzlich wurde eine weitere Sequenz aus Rhodoplasten von
von Corethron criophilum hinzugefügt.
Diesmal gab es 1393 alignierte Spalten im Alignment, von denen
730 unterschiedliche Spalten durch pfastDNAml
zur Stammbaumrekonstruktion verwendet wurden.
Solche Veränderungen der Anzahl der alignierten Spalten ergeben
sich daher, daß die verschiedenen rRNA-Sequenzen an den Enden
länger oder kürzer sind als andere.
Aus den Basenfrequenzen 0,266 (A), 0,219 (C), 0,305 (G) und 0,210 (T)
wurde als Transition/Transversions-Parameter 1,455 berechnet.
Nach sechs globalen Rearrangements (je 4290 Bäume) und insgesamt
28526 untersuchten Bäumen wurde einer mit einem
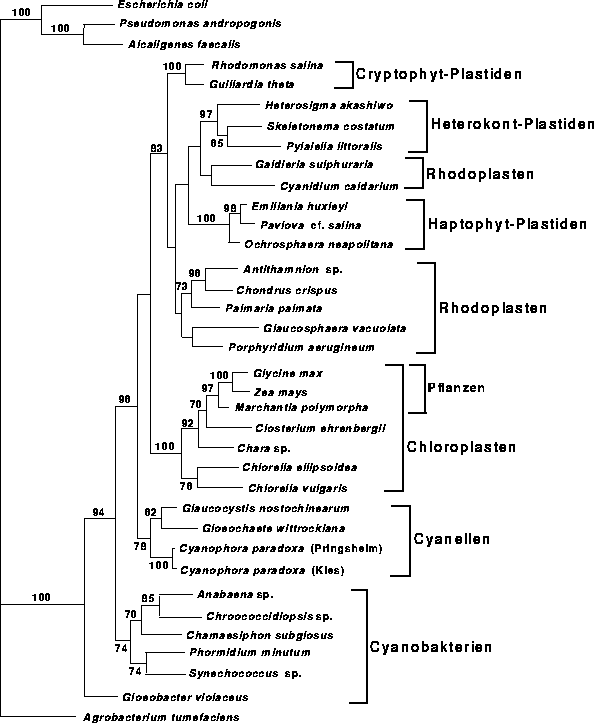
Log-Likelihood-Wert von -18016,00 gefunden (Abb. 6.12).
Abbildung:
Phylogenetischer Stammbaum der Plastidenentwicklung;
konstruiert
aus 16S rRNA-Sequenzen (Datensatz plast2 ) mittels pfastDNAml;
nur Bootstrap-Werte über 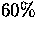 wurden an den
entsprechenden Kanten eingefügt
wurden an den
entsprechenden Kanten eingefügt
 |
Anhand dieses Datensatzes wurde eine Bootstrap-Analyse mit
100 gezogenen Pseudostichproben durchgeführt.
Die Bootstrap-Werte, die 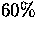 überschreiten, sind auch in
Abb. 6.12 dargestellt.
überschreiten, sind auch in
Abb. 6.12 dargestellt.



Next: Analyse mit gewichtetem Datensatz
Up: Datensatz plast2
Previous: Datensatz plast2
Heiko Schmidt
7/17/1997
![]() überschreiten, sind auch in
Abb. 6.12 dargestellt.
überschreiten, sind auch in
Abb. 6.12 dargestellt.