


Next: Analyse mit allen Sequenzstellen
Up: Aktinentwicklung
Previous: Aktinentwicklung
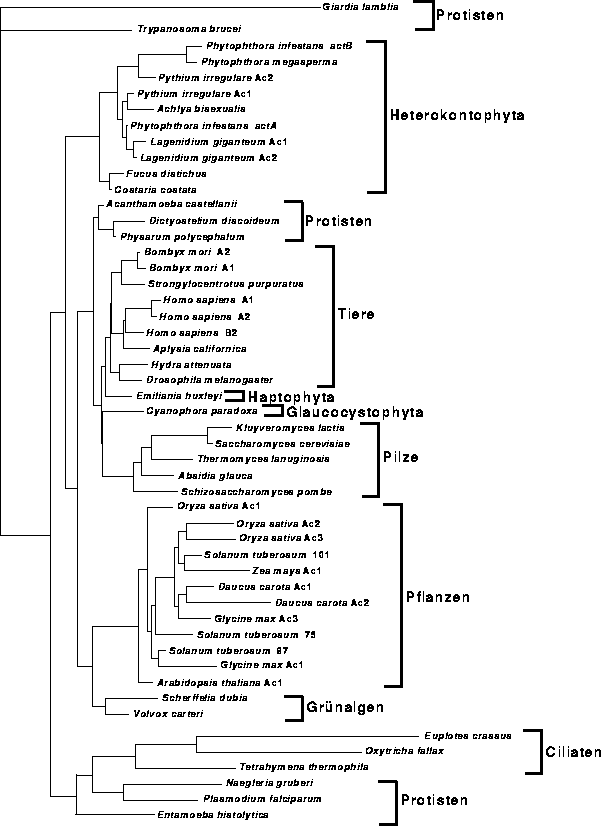
Abbildung:
Phylogenetischer Stammbaum der Aktinentwicklung;
berechnet mit pfastDNAml unter Benutzung der
Codon-Positionen 1 und 2
 |
Da die dritte Base der Codons der Aktingene sehr variabel
ist, wird diese Base bei phylogenetischen Analysen normalerweise
außeracht gelassen.
Der G+C-Gehalt an dieser Position bewegt sich bei unterschiedlichen
Spezies zwischen 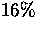 und
und 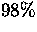 (z.B. Emiliania Huxleyi
(z.B. Emiliania Huxleyi 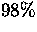 , Entamoeba histolytica
, Entamoeba histolytica 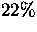 , Volvox carteri
, Volvox carteri 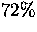 ).
Dagegen beträgt der G+C-Gehalt der anderen
Positionen
).
Dagegen beträgt der G+C-Gehalt der anderen
Positionen 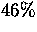 -
-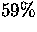 (erste Position) bzw.
(erste Position) bzw.
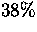 -
-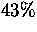 (zweite Position).
Diese Eigenschaften hängen auch nicht mit den einzelnen Gruppen
von Spezies zusammen, sondern variieren auch innerhalb der
verschiedenen Gruppen.
(Bhattacharya and Ehlting, 1995; Bhattacharya et al., 1993)
(zweite Position).
Diese Eigenschaften hängen auch nicht mit den einzelnen Gruppen
von Spezies zusammen, sondern variieren auch innerhalb der
verschiedenen Gruppen.
(Bhattacharya and Ehlting, 1995; Bhattacharya et al., 1993)
Aus diesem Grund wurde auch hier ein Stammbaum mit nur den ersten
beiden Codon-Positionen der Sequenz konstruiert.
Der Datensatz bestand hier aus 51 Spezies mit einem Alignment aus
702 Basen. pfastDNAml benutzte hiervon 485 unterschiedliche
Spalten.
Anhand der empirisch errechneten Basenfrequenzen 0,297 (A), 0,222 (C),
0,254 (G) und 0,227 (T) wurde ein Transition/Transversions-Parameter
von 1,4807 bestimmt. Es wurden bei der Berechnung des Baumes 96823
verschiedene Bäume betrachtet und 10 globale Rearrangements
ausgeführt. Der so gefundene Baum (Abb. 6.1)
hat einen Log-Likelihood-Wert
von -10585,69.



Next: Analyse mit allen Sequenzstellen
Up: Aktinentwicklung
Previous: Aktinentwicklung
Heiko Schmidt
7/17/1997

