


Next: Bootstrap-Verfahren
Up: Methoden, Modelle und Algorithmen
Previous: Suche nach dem optimalen
Um eventuelle unerwünschte Effekte zu vermeiden,
die durch hochvariable Bereiche in
den Eingabesequenzen auftreten können, wurden teilweise gewichtete
Datensätze erstellt.
Gesucht wurden Gewichte für die einzelnen Spalten im Sequenzalignment,
so daß hochvariable Spalten heruntergewichtet werden, während
konserviertere Basen im Alignment ein höheres Gewicht erhalten.
Dazu wurde die Möglichkeit des Programms MacClade genutzt,
Weightsets zu generieren (Maddison and Maddison, 1992).
Dem Programm wurde ein ML-Stammbaum
als Grundlage für die Gewichtung übergeben.
MacClade generiert dann nach der Parsimony-Methode
Sequenzen für die internen Knoten.
Dieses geschieht so, daß möglichst wenig Mutationen für die
Realisierung des Baumes nötig sind (siehe Kap. 1.4.3).
Anschließend werden die notwendigen Mutationen in jeder Alignmentspalte
gezählt.
Die Anzahl der Mutationen S (Parsimony-Steps) ist die Grundlage
zur Berechnung der Gewichte.
Als Gewichtungsfunktion W wurde
| 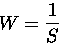 |
(10) |
verwendet. Durch Verwendung der Option integral weights
werden die einzelnen Werte W mit der gewünschen Anzahl an
Gewichten multipliziert und anschließend auf den nächsten ganzzahligen
Wert gerundet. Dieser Wert entspricht dem errechneten Gewicht.
Spalten mit S=0, d.h. ohne Mutationen, erhalten ebenfalls das geringste
Gewicht, da sie für die Stammbaumanalysen keine Aussagekraft besitzen.
Es wurden im allgemeinen 10 Gewichte gewählt, die
später bei der Analyse mit pfastDNAml jeweils
einer Gewichtskategorie zugeordnet wurden.



Next: Bootstrap-Verfahren
Up: Methoden, Modelle und Algorithmen
Previous: Suche nach dem optimalen
Heiko Schmidt
7/17/1997