


Next: Parallelisierung und Laufzeituntersuchung
Up: Methoden, Modelle und Algorithmen
Previous: Erstellen von gewichteten Datensätzen
Das in dieser Arbeit verwendetes Verfahren zum
Testen der Aussagekraft der errechneten
Bäume ist das von Efron (1979) entwickelte Bootstrap-Verfahren.
Das Bootstrap-Verfahren wird verwendet, um aus den vorhandenen
Daten neue unabhängige Stichproben zu gewinnen (sog. Pseudo-Stichproben).
Dies geschieht durch zufälliges Ziehen neuer Stichproben aus einer
vorhandenen. Es wird so lange gezogen, bis wieder die Gesamtzahl
an Einzelstichproben erreicht ist.
Dabei werden einige Einzelstichproben aus der Originalstichprobe
nicht mehr, andere dafür mehrfach in der Pseudostichprobe
vorhanden sein.
Diese Methode wurde von Neyman (1971) zum Testen
phylogenetischer Stammbäume vorgeschlagen und von
Felsenstein (1985) umgesetzt.
Hierbei werden die Organismen konstant gehalten, während
die Alignmentspalten neu gezogen werden.
Zum Gewinnen von Bootstrap-Stichproben wurden zwei Verfahren
angewendet:
- 1.
- Erstellen von Bootstrap-Stichproben durch
das PHYLIP-Programm seqboot (Felsenstein, 1993)
- 2.
- Nutzung der Bootstrap-Option B innerhalb von
pfastDNAml und damit verbunden die Angabe
eines Bootstrap-Random-Number-Seed zur
Berechnung einer Reihe von Zufallszahlen.
Es wurden immer 100 unabhängige Bootstrap-Stichproben
generiert.
Hundert Bootstrap-Stichproben sind nach Hedges (1992)
die minimale Anzahl, die
nötig ist, um einen aussagekräftigen P-Wert von 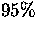 erhalten zu können.
erhalten zu können.
Zur Auswertung dieser 100 Stichproben wurde das PHYLIP-Programm
consense verwendet (Felsenstein, 1993), das aus den errechneten
Bäumen einen Konsensusbaum nach dem Majoritätsprinzip
generiert und zu jedem Teilbaum den Bootstrap-Wert angibt,
mit welcher prozentualen Häufigkeit dieser Teilbaum in allen
Bäumen gefunden wurde.



Next: Parallelisierung und Laufzeituntersuchung
Up: Methoden, Modelle und Algorithmen
Previous: Erstellen von gewichteten Datensätzen
Heiko Schmidt
7/17/1997