 |
 |
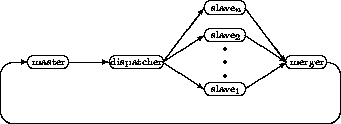
Vergleicht man das gewählte Parallelisierungskonzept (Abb. 5.1) mit dem, das bei der früheren P4-Implementierung von fastDNAml verwendet wurde (Abb. 5.7, nach Matsuda et al. 1994, unveröffentlicht), so fällt auf, daß bei pfastDNAml weit weniger verschiedene Prozeßtypen implementiert wurden. In fastDNAml gab es neben den Master- und Slave-Prozessen noch den Dispatcher-Prozeß, der das Verteilen der Bäume an die Slave-Prozesse übernimmt, und einen Merger-Prozeß, der die fertigen Bäume empfängt und den besten an den Master-Prozeß übermittelt. Außerdem war noch ein, in der Abbildung nicht gezeigter, Host-Prozeß implementiert, der die Initialisierung des Gesamtprozesses übernahm.
Die Aufgaben des Dispatcher-, Merger- und Host-Prozesses werden bei pfastDNAml durch den Master-Prozeß mitübernommen. Untersuchungen bei laufender Berechnung haben ergeben, daß der Master-Prozeß von pfastDNAml viel Zeit damit verbringt, darauf zu warten, daß er die schon fertig generierten Bäume an die Slave-Prozesse übermitteln kann. Da der Master-Prozeß also nicht in seiner Hauptaufgabe, dem Generieren der Bäume, gestört war, bestand bei pfastDNAml nicht die Notwendigkeit, die Kommunikation mit den Slave-Prozessen auszulagern. Die hierdurch gesparten drei Knoten können bei pfastDNAml-Analysen gut durch entsprechend mehr Slave-Prozesse genutzt werden, die sich, wie oben angesprochen, bei größeren Analysen positiv auf die Laufzeit auswirken.